что такое днк полимераза
ДНК-полимеразы
ДНК-полимераза — фермент, участвующий в репликации ДНК. Ферменты этого класса катализируют полимеризацию дезоксирибонуклеотидов вдоль цепочки нуклеотидов ДНК, которую фермент «читает» и использует в качестве шаблона. Тип нового нуклеотида определяется по принципу комплементарности с шаблоном, с которого ведётся считывание. Собираемая молекула комплементарна шаблонной моноспирали и идентична второму компоненту двойной спирали.
Выделяют ДНК-зависимую ДНК-полимеразу (КФ 2.7.7.7), использующую в качестве матрицы одну из цепей ДНК, и РНК-зависимую ДНК-полимеразу (другое название обратная транскриптаза, КФ 2.7.7.49), способную также к считыванию информации с РНК (обратная транскрипция).
ДНК-полимеразу считают холоферментом, поскольку для нормального функционирования она требует присутствия ионов магния в качестве кофактора. В отсутствии ионов магния о ней можно говорить как об апоферментe.
ДНК-полимераза начинает репликацию ДНК, связываясь с отрезком цепи нуклеотидов. Среднее количество нуклеотидов, присоединяемое ферментов ДНК-полимеразой за один акт связывания/диссоциации с матрицей, называют процессивностью.
Содержание
Действие ДНК-полимеразы
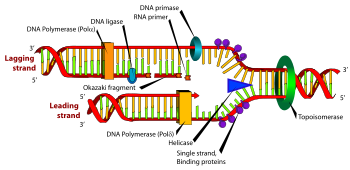
ДНК-полимераза добавляет свободные нуклеотиды к 3’-концу собираемой цепочки. Это приводит к элонгации цепочки в направлении 5′-3′. Ни одна из известных ДНК-полимераз не может создать цепочку «с нуля»: они в состоянии лишь добавлять нуклеотиды к уже существующей 3’-гидроксильной группе. По этой причине ДНК-полимераза нуждается в праймере, к которому она могла бы добавить первый нуклеотид. Праймеры состоят из оснований РНК и ДНК, при этом первые два основания всегда РНК-основания. Праймеры синтезируются другим ферментом — праймазой. Еще один фермент — геликаза — необходим для раскручивания двойной спирали ДНК с формированием одноцепочечной структуры, которая обеспечивает репликацию обеих цепочек в соответствии с полуконсервативной моделью репликации ДНК.
Некоторые ДНК-полимеразы обладают также способностью исправлять ошибки во вновь собираемой цепочке ДНК. Если происходит обнаружение неправильной пары нуклеотидов, ДНК-полимераза откатывается на один шаг назад. Благодаря своему экзонуклеазному действию ДНК-полимераза может исключить неправильный нуклеотид из цепочки и затем вставить на его место правильный, после чего репликация продолжается в нормальном режиме.
Многообразие ДНК-полимераз
Структура ДНК-полимераз достаточно жестко фиксирована. Их каталитические субъединицы очень мало различаются в различных видах живых клеток. Такая фиксация структуры обычно появляется там, где отсутствие разнообразия обусловлено огромной важностью или даже незаменимостью для функционирования клетки.
Генами некоторых вирусов тоже кодируются особые ДНК-полимеразы, которые могут избирательно реплицировать вирусные ДНК. Ретровирусы обладают геном необычной ДНК-полимеразы, называемой еще обратной транскриптазой, являющейся РНК-зависимой ДНК-полимеразой и осуществляющей сборку ДНК на основе шаблонной РНК.
Семейства ДНК-полимераз
На основании своей структуры ДНК-полимеразы могут быть разбиты на семь различных семейств: A, B, C, D, X, Y, и RT.
Семейство A
Семейство A включает в себя репликативные и восстановительные ДНК-полимеразы. Репликативные члены этого семейства представлены, например, хорошо исследованной ДНК-полимеразой вируса Т7 или эукариотической митохондриальной ДНК-полимеразой γ. Среди восстановительных полимераз мы находим такие примеры как ДНК-полимераза I E. coli, полимераза I из Thermus aquaticus или полимераза I Bacillus stearothermophilus. Восстановительные полимеразы участвуют в процессе устранения ошибок в собираемой ДНК, а также в обработке фрагментов Оказаки.
Семейство B
В семейство B в основном входят восстановительные полимеразы, в том числе основные эукариотические ДНК-полимеразы α, δ, и ε, а также ДНК-полимераза ζ. К этому семейству также относят ДНК-полимеразы некоторых бактерий и бактериофагов, например бактериофагов T4, Phi29 and RB69. Эти ферменты используются в синтезе и 3’-5’, и 5’-3’ моноцепей ДНК. Отличительной особенностью полимераз этого семейства является замечательная точность репликации. Многие также обладает сильным 3’-5’-экзонуклеазным действием (за исключением ДНК-полимераз α и ζ, у которых способности корректировать ошибки не наблюдается).
Семейство C
Полимеразы этого семейства — в основном бактериальные хромосомные репликативные ферменты, обладающие, кроме того, 3’-5’-экзонуклеазным действием.
Семейство D
Полимеразы этого семейства недостаточно изучены. Все известные образцы считаются репликативными полимеразами и обнаружены у архей субдомена Euryarchaeota.
Семейство X
К семейству Х относится широко известная эукариотическая ДНК-полимераза β, а также и другие, такие как σ, λ, μ и концевая дезоксинуклеотидил-трансфераза (TdT). ДНК-полимераза β необходимого для осуществления процесса восстановления поврежденных участков ДНК. Полимеразы λ и μ участвуют в негомологическом соединении — процессе восстановления разрывов двойной спирали.. TdT экспрессируется только в лимфоидной ткани и добавляет «n нуклеотидов» к разрывам двойной спирали, образующимся во время В(Р)С-рекомбинации. Дрожжи Saccharomyces cerevisiae обладают лишь одной полимеразой X, Pol4, участвующей в негомологическом соединении.
Семейство Y
Полимеразы этого семейства отличаются от прочих низкой производительностью на целостных шаблонах, а также способностью осуществлять репликацию на шаблонах поврежденных ДНК. Вследствие этого члены семейства называются полимеразами транслезионного синтеза. В зависимости от характера повреждения (лезии) ТЛС-полимеразы могут восстановить исходную цепочку. Ошибка может и не быть восстановлена, что приводит к мутациям. Страдающие Xeroderma pigmentosum, например, обладают мутантным геном ДНК-полимеразы η (eta), который толерантен к повреждениям, однако другие полимеразы, например ζ (относящаяся к семейству B), страдают от мутациям, что, как считается, приводит к предрасположенности к онкологическим заболеваниям.
Другие члены этого семейства — человеческие полимеразы ι, κ, а также концевая дезоксинуклеотидил-трансфераза Rev1. У E.coli имеются две ТЛС-полимеразы: IV (DINB) и V (UMUC).
Семейство RT
Семейство обратных транскриптаз (название семейства происходит от англ. reverse transcriptase) содержит полимеразы, обнаруженные как у ретровирусов, так и у эукариот. Они являются РНК-зависимыми ДНК-полимеразами, то есть, в отличие от описанных выше ферментов, используют в качестве матрицы для синтеза РНК, а не ДНК. Эукариотические полимеразы в основном представлены теломеразами. Эти полимеразы используют шаблонную РНК для синтеза цепочки ДНК.
Прокариотические ДНК-полимеразы
У бактерий обнаружено пять ДНК-полимераз:
Эукариотические ДНК-полимеразы
Эукариоты содержат по меньшей мере пятнадцать видов ДНК-полимераз [1] :
Обнаружены и другие эукариотические полимеразы.
Ни одна эукариотическая полимераза не может отщеплять праймеры, то есть не обладает 5’-3’-экзонуклеазным действием. Эту функцию выполняют другие ферменты. Только полимеразы, осуществляющие элонгацию (γ, δ и ε) обладают 3′-5′-экзонуклеазными свойствами.
Одна из человеческих полимераз способна проводить обратную транскрипцию

Рис. 1. Модели пространственной структуры комплекса из полимеразы тета (Polθ) с дуплексом ДНК/ДНК + дидезоксиГТФ (слева) и с дуплексом ДНК/РНК + дидезоксиГТФ (справа). Непосредственный участок взаимодействия белка с присоединяемым нуклеотидом дидезоксиГТФ (ddGTP) показан дополнительно во врезках. В каталитическом домене полимеразы выделяют субдомены «ладонь» (palm), «пальцы» (fingers) и «большой палец» (thumb). Рисунок из дополнительных материалов к обсуждаемой статье в Science Advances
Нуклеиновые кислоты — носители генетической информации у всех обитателей Земли — представлены молекулами ДНК и РНК. Принцип комплементарности позволяет строить новые цепочки нуклеиновых кислот на матрице уже существующих. В клетках эукариот есть ферменты, которые катализируют синтез цепочек ДНК на матрице ДНК (репликацию) или синтез РНК на матрице ДНК (транскрипцию). У некоторых вирусов, например у ВИЧ, имеются ферменты, катализирующие синтез цепочек ДНК на матрице РНК (обратную транскрипцию). Группа американских ученых выяснила, что такой же способностью обладает и один из ферментов наших собственных клеток — ДНК-полимераза тета. Ранее было известно, что полимераза тета, играющая важную роль в репарации повреждений ДНК, при репликации делает большое количество ошибок, вставляя некомплементарные нуклеотиды. Теперь же выяснилось, что этот фермент охотно строит ДНК на матрице РНК, и к тому же ошибки в ходе такого синтеза появляются гораздо реже. По скорости синтеза ДНК на матрице РНК и по частоте ошибок при таком синтезе полимераза тета оказалась сопоставима с обратной транскриптазой ВИЧ.
В живых клетках ДНК — молекула генетической памяти — постоянно подвергается риску появления разнообразных повреждений. Само собой, это означает необходимость регулярного обслуживания по ремонту молекулы ДНК — этот процесс называется репарацией. Для репарации клетки располагают обширным набором механизмов и ферментов — выбор определяется типом повреждений, их количеством, стадией клеточного цикла и еще рядом факторов. В 2015 году за изучение систем репарации ДНК была присуждена Нобелевская премия (см. новость Нобелевская премия по химии — 2015, «Элементы», 09.10.2015).
Полимераза тета (DNA polymerase theta, Polθ) — один из ферментов системы репарации, работающий в клетках человека и других млекопитающих. Его назначение — спасти клетку от гибели в тех условиях, когда повреждений очень много и другие системы репарации не справляются (в первую очередь это касается повреждений типа 2-цепочечных разрывов ДНК). В наших клетках эта полимераза кодируется геном POLQ. Давно известная особенность Polθ — очень низкая точность синтеза ДНК. Именно эта особенность и позволяет ему беспрепятственно пройти через «трудные» места в ДНК, содержащие множество повреждений. Более точно работающие полимеразы в таких местах просто останавливаются, не будучи способными вставить какой-то нуклеотид напротив места, содержащего повреждение. Однако за использование фермента с низкой точностью становится приходится платить — в ДНК появляется много мутаций (главным образом замен) в нуклеотидной последовательности.
Интерес ученых к Polθ обусловлен в значительной мере тем фактом, что эта полимераза гораздо активнее работает в раковых клетках, чем в здоровых клетках организма. И это одна из причин, по которой раковые клетки очень быстро мутируют и «эволюционируют» внутри организма, приобретая выгодные для себя мутации (например, делающие их более устойчивыми к воздействию противораковых лекарств). Ингибирование этого фермента даже рассматривают как одну из перспективных стратегий борьбы с проблемой развития лекарственной устойчивости в ходе лечения онкологических пациентов (T. Kent et al., 2016. DNA polymerase θ specializes in incorporating synthetic expanded-size (xDNA) nucleotides).
Эволюционно Polθ происходит от Pol I — фермента, имеющегося у большинства прокариот, который обычно обеспечивает очень точную репликацию ДНК. В структуре Polθ, как и в структуре Pol I, имеется собственно каталитический домен и домен «самоконтроля» (proofreading), который позволяет высокоточной полимеразе, в случае вставки ошибочного нуклеотида, остановиться, вырезать неправильный нуклеотид позади себя и заменить его на другой. Благодаря этому домену полимераза не двинется дальше, пока не будет встроен верный нуклеотид. Однако в Polθ этот домен в ходе эволюции оказался подпорчен несколькими мутациями, из-за чего он перестал обеспечивать такой самоконтроль.
ДНК-полимеразы бывают разные. Однако все их можно подразделить на две большие группы — в зависимости от типа субстрата. Существуют ДНК-зависимые ДНК-полимеразы — таковыми являются все ранее известные ферменты, задействованные в репликации и репарации у клеточных организмов. Из названия следует, что эти ферменты строят новые цепочки ДНК на матрице родительских цепочек ДНК. Другую группу составляют РНК-зависимые ДНК-полимеразы (их же часто называют обратными транскриптазами) — они строят цепочки ДНК, используя в качестве матриц цепочки РНК. Такие ферменты есть у некоторых вирусов и у некоторых транспозонов — геномных паразитов, имеющих, предположительно, вирусное происхождение. Вопрос о том, выполняют ли обратные транскриптазы транспозонов какие-либо полезные функции для клетки является предметом дискуссий и мы его здесь касаться не будем, потому что нас в данном случае интересует совершенно родной белок клеток животных, не имеющий признаков вирусного происхождения. Однако кое в чем он оказался схож с полимеразами вирусов, поломав такую стройную картину классификации ДНК-полимераз по субстрату, которую мы только что представили.
Главное отличие между РНК и ДНК состоит в том, что в качестве сахара — обязательного компонента нуклеиновых кислот — в первом случае используется рибоза, во втором — дезоксирибоза. В сущности, вся разница сводится к наличию или отсутствию в молекуле сахара одного атома кислорода. Однако эта разница достаточно существенна, чтобы обеспечивать ферментам высокую специфичность при выборе субстрата при осуществлении матричного синтеза ДНК — это было показано для всех ранее изучавшихся ДНК-полимераз.
Наличие неактивного домена самоконтроля натолкнуло ученых из США на гипотезу, что, возможно, Polθ способна синтезировать ДНК не только на матрице ДНК, но и на матрице РНК. Для проверки этой гипотезы и было затеяно исследование, о котором пойдет речь. Параллельные испытания провели для обратной транскриптазы ВИЧ (синтез ДНК на матрице РНК — основная профессия обратной транскриптазы) и для еще одного человеческого фермента репарации, также склонного к ошибкам, — полимеразе эта (Polη).
В первом испытании ферментам дали наращивать цепочку ДНК на матрице РНК длиной 22 нуклеотида, с которой был связан ДНК-праймер длиной 11 нуклеотидов. Результаты можно видеть на рис. 2. Пятнышки, которые мы видим на фотографии электрофорезной пластинки — это фрагменты нарощенной ДНК. Если к праймеру присоединился один нуклеотид — получаем фрагмент длиной 12, нуклеотидов, если два — длиной 13 и т. д.

Рис. 2. Оценка способности разных полимераз наращивать цепочку ДНК по матрице РНК. А — накопление продуктов реакции полимеризации с течением времени. Показаны фотографии гелей, в которых электрофорезом разделялись продукты реакции. Левое фото — при посредстве обратной транскриптазы ВИЧ HIV RT, среднее — при посредстве полимеразы тета Polθ, правое — при посредстве полимеразы эта Polη. Над фотографиями схематично изображен начальный субстрат — дуплекс из РНК-матрицы (цепочка РНК показана красным цветом) и ДНК-затравки (цепочка ДНК показана черным цветом). Затравка имеет длину 11 нуклеотидов, так что пятна в геле, напротив которых стоит отметка 11 nt — это оставшиеся не удлиненными затравки. Цепочка матрицы имеет длину 22 нуклеотида — и это теоретически максимальная возможная длина продукта матричного синтеза ДНК (в геле видны и продукты несколько большей длины — это следствие способности полимераз добавлять нуклеотид нематричным способом). Цепочки промежуточной длины — это продукты незавершенной реакции полимеризации. Можно убедиться, что эффективный синтез ДНК идет только при работе ферментов HIV RT и Polθ. B доля затравок, удлиненных в течение указанного времени при работе HIV RT и Polθ. Видно, что динамика работы этих двух ферментов очень схожа. Рисунок из обсуждаемой статьи в Science Advances
Как видно, чем больше времени давалось ферменту (до 32 минут в общей сложности), тем больше получалось длинных фрагментов (в этой системе максимально возможными были фрагменты длиной 22 нуклеотида) — причем, только если в качестве фермента выступали Polθ или обратная транскриптаза ВИЧ (HIV RT). А вот фермент Polη в тех же концентрациях и за то же время успевал добавить не больше трех (правда при очень сильном повышении концентрации этого фермента удавалось добиться получения более протяженных фрагментов). Дополнительно ученые показали, что способность синтезировать ДНК на матрице РНК у Polθ (а также и у HIV RT) сохраняется при достаточно широком диапазоне условий (разных значениях pH и концентрации KCl). Полимераза Polη теряла такую способность при малейшем отклонении условий от оптимальных значений. Ни у одной другой ДНК-полимеразы человека (а проверили все восемь имеющихся у человека ДНК-полимераз) этой способности не выявлялось вовсе.
Ученые также показали и субстратную специфичность обратной транскриптазы ВИЧ — она могла эффективно работать только на матрице РНК, но с большим трудом строила фрагменты на матрице ДНК (длинные фрагменты получались только при очень высоких концентрациях фермента). А вот Polθ действительно эффективно работала и на матрице ДНК, и на матрице РНК.
Следующая серия экспериментов показала, что, во-первых, Polθ обладает более высоким сродством к гибридным молекулам ДНК/РНК, чем к ДНК/ДНК, во-вторых, синтез ДНК на матрице РНК идет и быстрее, и точнее, чем на матрице ДНК. Частично результаты этих экспериментов показаны на рис. 3.

Рис. 3. Скорость и точность синтеза полимеразой тета ДНК на матрицах РНК или ДНК. А — варианты субстратов, использованных в экспериментах — дуплексы, образованные ДНК-матрицей с ДНК-затравкой или РНК-матрицей с ДНК-затравкой (последовательности ДНК показаны черным шрифтом, последовательности РНК — красным). B — результаты серии экспериментов по оценке скорости включения нуклеотидов в ходе полимеризации ДНК. В ходе эксперимента в смесь помимо субстрата и фермента добавлялись меченные дезоксинуклеотиды только одного типа из четырех существующих (dCTP, dTTP, dATP или dGTP). Это был нуклеотид, комплементарный следующему за затравкой в матрице. Оценивали, какая доля затравок оказывалась помечена включением нуклеотида (% extension) в течение определенного промежутка времени (измеряемого в минутах, min). На всех графиках черные точки соответствуют экспериментам с ДНК-матрицей, а точки другого цвета — экспериментам с РНК-матрицей. Как можно заметить, скорость включения трех из четырех нуклеотидов была выше при работе на матрице РНК. C — результаты серии экспериментов по оценке точности включения нуклеотидов в ходе полимеризации ДНК. В реакционную смесь добавляли субстрат, фермент и меченные дезоксинуклеотиды единственного типа. Но теперь, наоборот, это был нуклеотид НЕ комплементарный следующему за затравкой в матрице: его включение обозначало ошибочное срабатывание фермента. Более высокой скорость удлинения затравки теперь оказалась для ДНК-матрицы, то есть фермент реже допускает ошибочное включение нуклеотида при работе на матрице РНК. Рисунок из обсуждаемой статьи в Science Advances
Вероятным объяснением различий в скорости и точности синтеза на ДНК- или РНК-матрицах является изменение конформации белка в зависимости от связываемого субстрата. Действительно, оказалось, что субдомены каталитического домена претерпевают достаточно заметные конформационные изменения при связывании дуплексов ДНК/РНК или ДНК/ДНК — это различие можно видеть на рис. 1. Оказалось, что несколько аминокислот в субдомене, именуемом «большой палец», образуют дополнительные водородные связи с рибозой при связывании гетеродуплекса ДНК/РНК, что, вероятно, и объясняет более высокое сродство фермента к субстрату такого типа, равно как и более высокую скорость и точность синтеза ДНК, чем при работе с субстратом типа ДНК/ДНК.
Все вышеописанные данные были получены при исследовании свойств белка вне живой системы — in vitro. Конечно, чтобы выводы были вполне убедительными, нужно было убедиться, что Polθ действительно может использовать РНК как матрицу, работая непосредственно в живой клетке в качестве починщика ДНК. Эксперименты оказались вполне успешными. Мы опишем лишь некоторые из них, выполненные на клетках человека.
Ученые работали с линией клеток остеосаркомы (опухоли кости) человека U2OS. В эти клетки был встроен репортерный ген GFP (кодирующий зеленый флуоресцентный белок). Работу такого гена можно видеть по свечению клеток. Однако в данном случае ген был «подпорчен» делецией семи нуклеотидов в середине последовательности, да еще и вставкой небольшого постороннего фрагмента сразу около делеции. Вторую тестовую линию — POLQ e16m — получили на основе вышеописанной, добавив еще одну модификацию — в этой линии полностью вывели из строя ген Polθ.
Первый эксперимент состоял в том, что в клетки родительской линии U2OS с дефектным GFP вносили небольшую цепочку нуклеиновой кислоты, содержащий верную последовательность центральной части гена GFP — без инсерции и с возвращенными семью нуклеотидами. Этот фрагмент должен был служить матрицей для заделывания бреши в гене GFP. Для того, чтобы сделать брешь и запустить механизм репарации, использовали систему CRISPR/Cas, которую натравили на встроенный в ген GFP чужеродный участок.
Нуклеиновая кислота, которая должна была служить матрицей для починки хромосомы, представляла собой либо целиком одноцепочечную молекулу ДНК, либо содержала два рибонуклеотида, попадающие в те самые недостающие семь нуклеотидов внутри GFP. Свечение клеток в итоге могло появиться только в том случае, если при заполнении бреши будет верно «списана» последовательность соответствующей цепочки фрагмента для редактирования. Рис. 4 показывает, что свечение восстанавливалось в большинстве клеток при использовании как цепочки, полностью построенной из дезоксирибонуклеотидов, так и при использовании цепочки с вставкой из двух рибонуклеотидов. Если тот же эксперимент проделывали с линией POLQe16m, то восстановления свечения при введении цепочки с рибонуклеотидами не наблюдалось, а вот если в эту линию вносили плазмиду с геном Polθ, то возвращалась и способность чинить хромосому. Схема и результаты этих серий экспериментов показаны на рис. 4.

Присутствие в геномной ДНК участков, образованных гетеродуплексами ДНК/РНК было установлено достаточно давно. Это могут быть единичные встроенные рибонуклеотиды, а могут быть участки с достаточно большой протяженностью. Причины их образования, как и механизмы их устранения, бывают разными — этому вопросу посвящен большой недавний обзор G. M. Nava et al., 2020. One, No One, and One Hundred Thousand: The Many Forms of Ribonucleotides in DNA. Это может быть естественной частью некоторых молекулярно-биологических процессов (рис. 5): транскрипции, репарации, репликации (в частности, из рибонуклеотидов строятся фрагменты Оказаки — затравки для репликации запаздывающей цепи). Вместе с тем рибонуклеотиды достаточно часто ошибочно включаются ферментами в ходе синтеза ДНК при репликации или репарации (приблизительная частота таких ошибок 10 −3 ), несмотря на высокую избирательность в пользу дезоксирибонуклеотидов. Это объясняется тем, что в наших клетках концентрация рибонуклеотидов всегда намного (до 100 раз) выше концентрации дезоксирибонуклеотидов. Впрочем, в клетках есть несколько видов охранных систем, которые настроены на выявление и удаление рибонуклеотидов из ДНК — и это очень важно для поддержания нормальной работы генов, репликации ДНК и сохранения ее в целости и сохранности.
Рис 5. Разнообразные механизмы появления дуплексов типа ДНК/РНК в хромосомном материале клеток. В клетках эукариот имеется обширный арсенал средств для устранения таких временно образующихся гетеродуплексов и восстановления нормальной структуры хромосом в форме двухцепочечной ДНК. Рисунок из обзорной статьи G. M. Nava et al., 2020. One, No One, and One Hundred Thousand: The Many Forms of Ribonucleotides in DNA
Но вот Polθ, исходя из результатов обсуждаемого исследования, обладает способностью использовать непосредственно рибонуклеотиды в качестве матрицы для синтеза ДНК. И эта ее способность пригождается на этапе репарации повреждений ДНК, по-видимому, главным образом тогда, когда по тем или иным причинам забуксовали другие охранные механизмы.
Таким образом, у млекопитающих впервые обнаружили фермент репарации с несколькими уникальными свойствами, включающими особенности структурной пластичности, способность прочно связывать гетеродуплексы ДНК/РНК и способность быстро и сравнительно с высокой точностью осуществлять обратную транскрипцию, выстраивая ДНК на матрице РНК. Это объясняет, каким образом наши клетки справляются с одним из достаточно частых нарушений в структуре ДНК, когда часть цепочки оказывается построена не из дезоксирибонуклеотидов, а из рибонуклеотидов.